Manual jp
Contents
Windows環境におけるpythonでの解析環境の構築マニュアル
注意:このマニュアルに書いてあるソフトウェアをインストールする際には、パソコンの管理者権限が必要です。また、windowsに関しては32bit版を想定しております。
Python
- http://www.python.org/download/releases/2.6.6/ から、Pythonのバージョン2.6.6の"Windows x86 MSI Installer"をダウンロード
- インストーラーを起動
- インストーラープログラムを以下の手順で進める
- 特に指定が無い画面については、"Next"ボタンを押して先に進めてください。
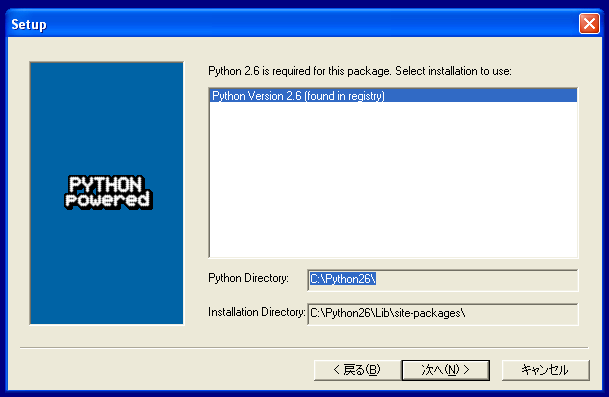
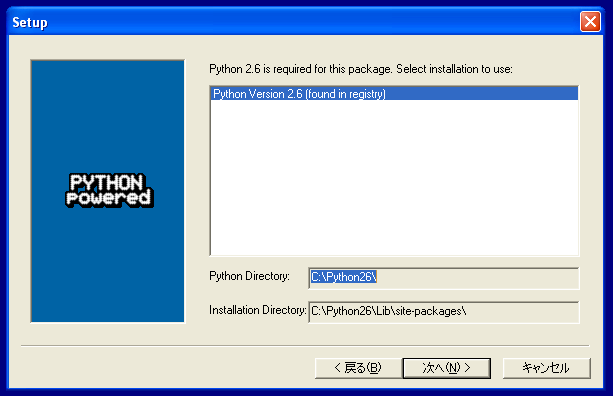
 "Install for all users"を選択して、"Next"ボタンを押す
"Install for all users"を選択して、"Next"ボタンを押す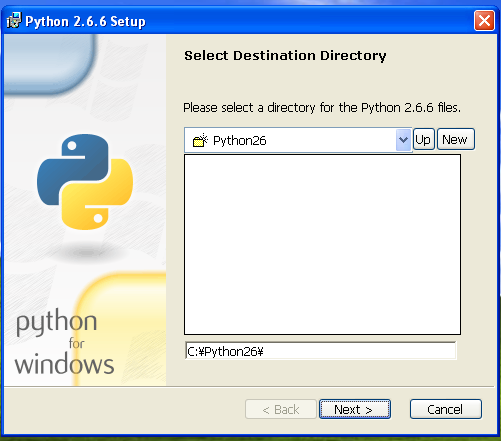 画面に書かれているものから変更せずにそのまま"Next"ボタンを押す
画面に書かれているものから変更せずにそのまま"Next"ボタンを押す- File:Data/050998d9a57e931e46c2c70119982d27.png "Next"ボタンを押してインストールを開始する
Numpy
Numpyはオープンソースのpython用の数値計算用のライブラリです。
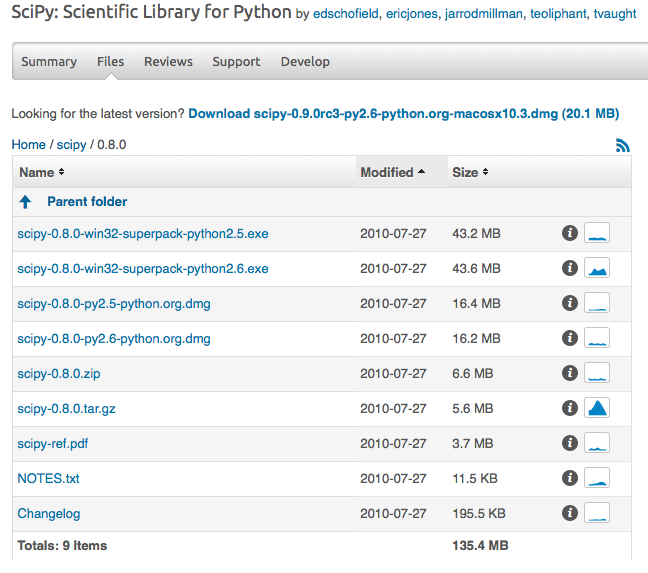
- http://sourceforge.net/projects/numpy/files/NumPy/1.5.1/ から、"numpy-1.5.1-win32-superpack-python2.6.exe"をダウンロード
- File:d9f9e4c26024b18dc4b21b099e7f900d.png]]
- インストーラーを起動
- インストーラープログラムを以下の手順で進める
Scipy
Scipyはオープンソースのpython用数学、科学、工学のための数値解析モジュールです。
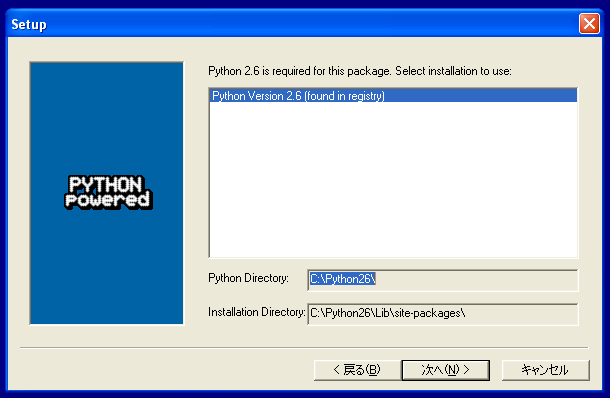
- http://sourceforge.net/projects/scipy/files/scipy/0.8.0/ から、"scipy-0.8.0-win32-superpack-python2.6.exe" をダウンロード
- インストーラーを起動
- インストーラープログラムを以下の手順で進める
Matplotlib
Matplotlibはpython用のグラフ描画の為のライブラリです。
- matplotlibのページ http://matplotlib.sourceforge.net/ にアクセス
- download リンクをクリックしてダウンロードページに移動.
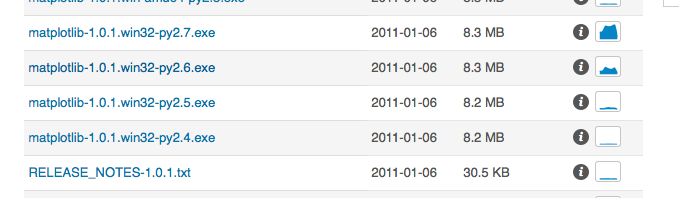
- "matplotlib-1.0.1.win32-py2.6.exe".をダウンロード
- インストーラーを起動
- インストーラープログラムを以下の手順で進める
R
Rはオープンソースの統計解析用のプログラムです。
- Rのページ http://www.r-project.org/ にアクセス
- ミラーを選択 http://cran.r-project.org/mirrors.html
- ここでは http://essrc.hyogo-u.ac.jp/cran/ を選択
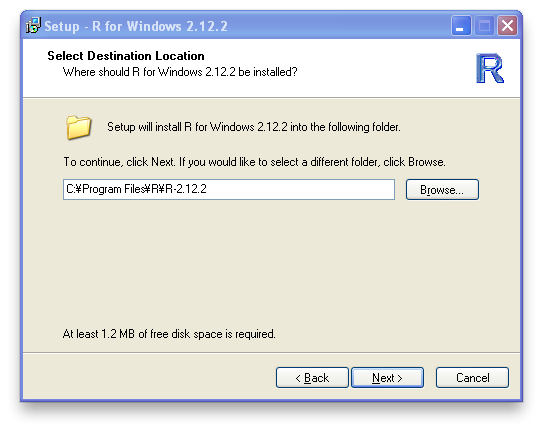
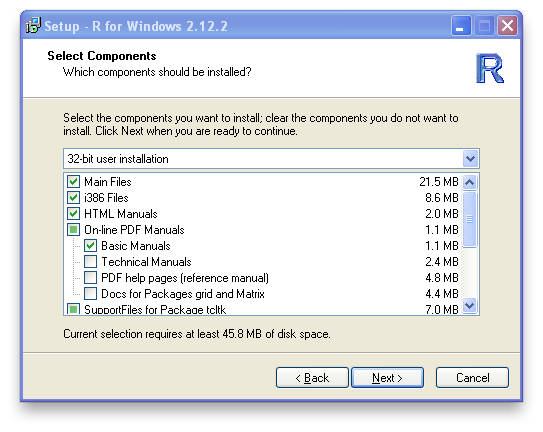
- リンクをWindows- > base とたどって win32用の最新バイナリ http://essrc.hyogo-u.ac.jp/cran/bin/windows/base/R-2.12.2-win.exe をダウンロード
- インストーラを起動
- インストーラプログラムを以下の手順で進める
rpy2
rpy2はpythonから統計解析システムRを利用するためのライブラリです
- rpy2のサイト http://sourceforge.net/projects/rpy/
- python2.6用のwindows用バイナリファイル http://sourceforge.net/projects/rpy/files/rpy2/2.0.x/rpy2-2.0.8.win32-py2.6.msi/download?use_mirror=jaist をダウンロード
- インストーラプログラムを以下の手順で進める
- 特に指定が無い画面については、"Next"ボタンを押して先に進めてください。
Pythonを用いてneutorychoのデータを扱うSample
neurotyhcoのデータは、"C:/neurotycho"以下に保存して解凍してあることを前提に進めます。別の場所に保存している場合は、そこを適宜読み替えて進めてください。
ECoGデータのプロット
Sleeping Taskのデータを対象に表示
#!/usr/bin/python
#-*- coding: utf-8 -*-
import numpy as np
import scipy.io
import scipy
import matplotlib
import matplotlib.pyplot as plt
_indir = "C:/neurotycho/20100604_S1_ST_K2_ToruYanagawa_mat_ECoG128-Event3/"
def load_mat(path):
"""
loading a matlab data using scipy.io
"""
return scipy.io.loadmat(path)
if __name__=="__main__":
mat = load_mat(_indir+"ECoG_ch1.mat")#loading ECoG data
data = mat["ECoGData_ch1"][0]
event = load_mat(_indir+"Event.mat")##loading event data
eindex = event["EventIndex"][0]#EventIndex is one-row vector contains indexes when events were occurred in ECoG data.
fig = plt.figure()
ax = fig.add_subplot(111)
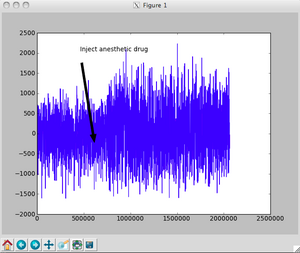
ax.plot(data)#plotting ECoGdata
ax.annotate('Inject anesthetic drug',
xy=(eindex[0],data[eindex[0]]),
xytext=(eindex[0]-170000,2000),
arrowprops=dict(facecolor='black', shrink=0.1,),
)
plt.show()
result
薬液注入後にECoG波形に明らかな変化があることが分かる。
ECoGデータの電極間の相関を求める
#!/usr/bin/python
#-*- coding: utf-8 -*-
import numpy as np
import scipy.io
import matplotlib
import matplotlib.pyplot as plt
import glob
import re
_indir = "C:/neurotycho/20100723S1_VGT_K2_KazuhitoTakenaka-ToruYanagawa_mat_ECoG128-Event3/"
def load_mat(path):
return scipy.io.loadmat(path)
def load_ecog_mats(path):
"""
特定のディレクトリ以下のECoGデータを全て読み込んで配列に変換
"""
pat = re.compile(r'ch(\d+)\.')
files = glob.glob(path+"*_ch*")
dic={}
for f in files:
result = pat.search(f)
if result:
id = result.group(1)
name = "ECoGData_ch"+id
dic[int(id)] = load_mat(f)[name][0]
ret = []
for k in sorted(dic.keys()):#電極の番号順に並び替え
ret.append(dic[k])
return ret
if __name__=="__main__":
data = load_ecog_mats(_indir)
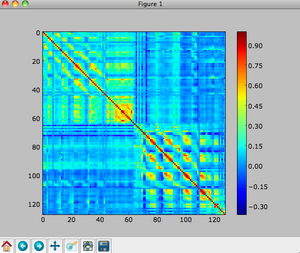
correlation_mat = np.corrcoef(data)#全実験期間中の電極間の相関を求める
fig = plt.figure()
ax = fig.add_subplot(111)
cax = ax.imshow(correlation_mat, interpolation='nearest')#二次元の画像として相関関係を描画
cbar = fig.colorbar(cax)
plt.show()
result
赤や黄色である電極同士は相関が高い。基本的には隣り合っている者どうしの相関が高くなっている。全てのデータでまとめて計算しているためこのデータには余り意味が無いが信号の相関の計算と描画方法のデモである。
刺激ごとの加算平均を求める
脳計測データは多くのノイズを含むため、加算平均を行って、意味のある信号を強化し、ノイズをうちけす必要がある。闇雲に加算するのではなく、同じ刺激を与えた場合の信号だけを、刺激提示のタイミングも揃えて加算する必要がある。
#!/usr/bin/python
#-*- coding: utf-8 -*-
import numpy as np
import scipy.io
import scipy
import matplotlib
import matplotlib.pyplot as plt
_indir = "C:/neurotycho/20100723S1_VGT_K2_KazuhitoTakenaka-ToruYanagawa_mat_ECoG128-Event3/"
def load_mat(path):
return scipy.io.loadmat(path)
def is_transit_state(laststate,nowstate,index,dic):
"""
状態が遷移したのかどうかを評価
"""
if laststate==nowstate:
return 0;
else:
if laststate==0:
dic[nowstate].append(index)
else:
dic[laststate].append(index-1)
def get_event(path):
"""
特定の傾きの画像をサルに提示を開始した時間と終了した時間のペアを作成
"""
event=load_mat(path+"Event.mat")["EventData"][0]
length = len(event)
state=0
dic=defaultdict(list)
for i in range(length):
signal = event[i]
if signal>=650 and signal<=750:
is_transit_state(state,1,i,dic)
state=1
elif signal>=950 and signal<=1050:
is_transit_state(state,2,i,dic)
state=2
elif signal>=1300 and signal<=1400:
is_transit_state(state,3,i,dic)
state=3
elif signal>=1600 and signal<=1700:
is_transit_state(state,4,i,dic)
state=4
elif signal>=1950 and signal<=2050:
is_transit_state(state,5,i,dic)
state=5
elif signal>=2250 and signal<=2350:
is_transit_state(state,6,i,dic)
state=6
elif signal>=2600 and signal<=2700:
is_transit_state(state,7,i,dic)
state=7
elif signal>=2900 and signal<=3000:
is_transit_state(state,8,i,dic)
state=8
else:
is_transit_state(state,0,i,dic)
state=0
for k,v in dic.iteritems():
dic[k]=zip(v[0::2],v[1::2])#提示開始時刻と提示終了時刻のペアを作成
return dic
def calc_avg_signal(id,event):
"""
傾きごとの実験期間のデータと、対象とする電極を与えて、傾きごとの加算平均信号を計算する
"""
ecog = load_mat(_indir+"ECoG_ch%i.mat" % (id))["ECoGData_ch%i"% (id)][0]
data = []
for j in range(8):
stimuli = event_dic["%i" % (j+1)]
ret = []
for pair in stimuli:
ret.append(ecog[pair[0]:pair[1]][0:1990])
data.append(sum(ret)/len(stimuli)+0.0)
return data
if __name__=="__main__":
event_dic = get_event(_indir)
id = 123
data = calc_avg_signal(id,event_dic)#123番目の電極における、刺激ごとの加算平均信号を計算
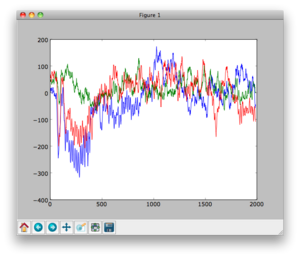
fig = plt.figure()
ax = fig.add_subplot(111)
ax.plot(data[1])#傾き90度の時の波形 pythonの配列は0から始まるので一つずれた値で指定する
ax.plot(data[3])#傾き180の時の波形
ax.plot(data[7])#傾き360の時の波形
plt.show()
result
青が90度、緑が180度、赤が360度の時の加算平均信号である。傾きに反応するV1視覚野は電極?であるので、その周辺の電極のデータの調査などが重要である。
Rとpythonの連携
Rを用いて、ECoGデータに対して短時間高速フーリエ変換を掛ける。
#!/usr/bin/env python
# -*- coding: utf-8 -*-
import numpy as np
import scipy.io
from rpy2 import robjects
import scipy
import matplotlib
import matplotlib.pyplot as plt
_indir = "C:/neurotycho/20100604S1_ST_K2_ToruYanagawa_mat_ECoG128-Event3/"
def load_mat(path):
return scipy.io.loadmat(path)
if __name__=="__main__":
try:
robjects.r["library"]("e1071")
pass
except:
robjects.r["options"](repos="http://cran.md.tsukuba.ac.jp")
robjects.r["install.packages"]("e1071")
robjects.r["library"]("e1071")
mat = load_mat(_indir + "ECoG_ch1.mat")["ECoGData_ch1"][0]
stft = robjects.r["stft"](robjects.FloatVector(mat))
robjects.r["png"]()
robjects.r["plot"](stft)
robjects.r["dev.off"]()
result
睡眠タスクのデータに対して短時間フーリエ変換を適用している。薬品注入前と注入後で、含まれる周波数特性が変わっているのが分かる。